В базе данных KEGG я выбрал метаболический путь Phenylalanine metabolism (метаболизм фенилаланина)
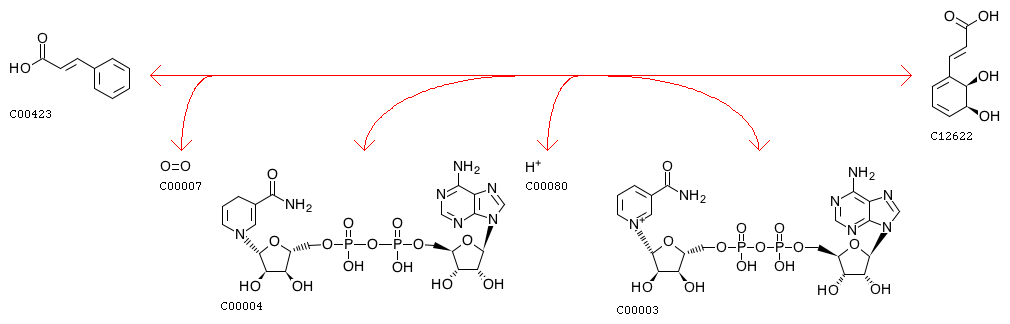
Затем я выбрал реакцию EC 14.12.19, которую катализирут четыре ортологических ряда белков(K00529, K05708, K05709, K05710)

Phenylpropanoate + Oxygen + NADH + H+ <=> cis-3-(Carboxy-ethyl)-3,5-cyclo-hexadiene-1,2-diol + NAD+
Второй и третий ряд являются субъединицами одного и того же фермента, поэтому я выбрал пути K00529 и K05708. Информация о них приведена в таблице.
| Информация об ортологичных рядах | ||
| Идентификатор | Число белковых последовательностей | Число генов |
| K05708 | 55 (47 из Uniprot) | 77 |
| K00529 | 466 (448 из Uniprot) | 1562 |
Так как ряд K00529 содержит слишком много белков, я возьму из него 57 последовательностей
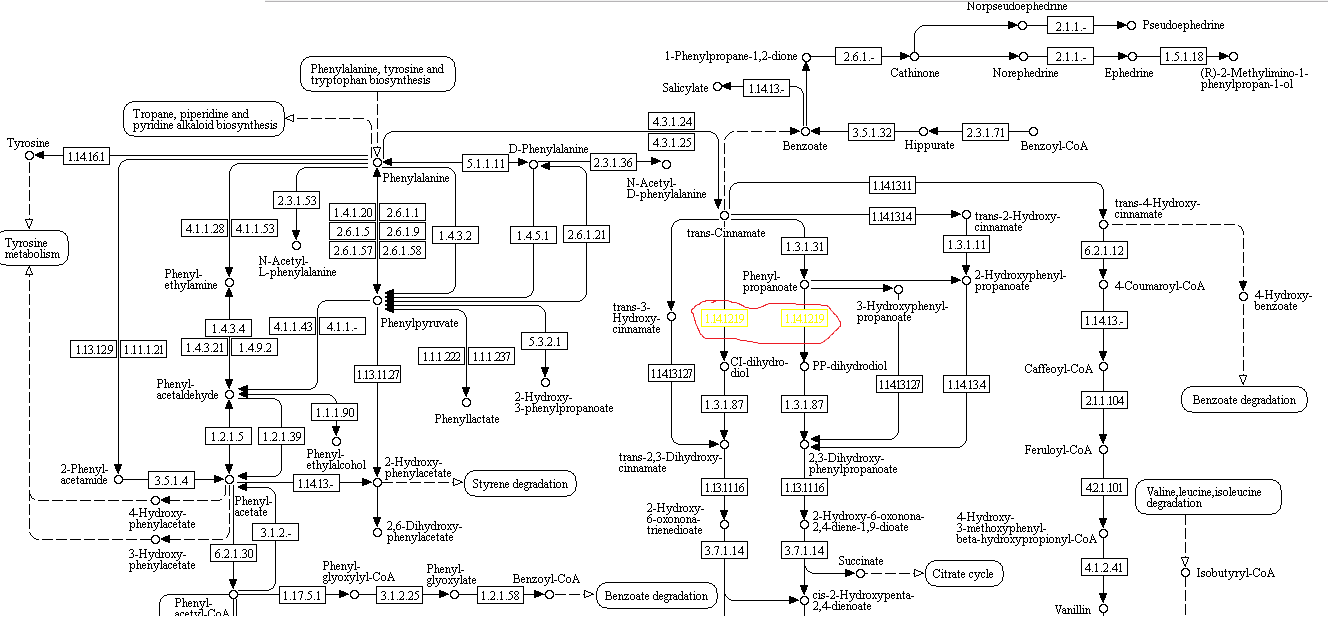
Картинка части метаболического пути с данной реакцией (обведена красным)
Файлы fasta-формата с белковыми последовательностями, которые будут использоваться для анализа:
При помощи программы MUSCLE получим множественное выравнивание наших последовательностей, которое затем было открыто в JalView и раскрашено по Clustalx
Последовательности сильно различаются по длине (примерно 830 аминокислот у K00529 и 500 у K05708). При этом присутствуют последовательности, у которых есть не встречающиеся у остальных участки(9MUCO, SPMWW, SMIFL, STRSW, FAHILE). Я удалил их и снова выровнял.
Короткие белки, которые содержат много гэпов в тех участках, где в других последовательностях выравнивания консервативные колонки, в моем выравнивании отсутствуют.
Новое множественное выравнивание
Можно видеть, что последовательности из разных рядов белков сильно отличаются. Гомология между белками одгого ряда присутствует. Можно увидеть, что последовательности образуют небольшие группы с наибольшим сходством внутри одного ряда.
Однако нельзя говорить о гомологии для двух разных рядов белков. Поэтому я считаю, что в данном случае бессмысленно строить дерево, так как оно будет иметь мало смысла.
© Борисов Евгений 2017